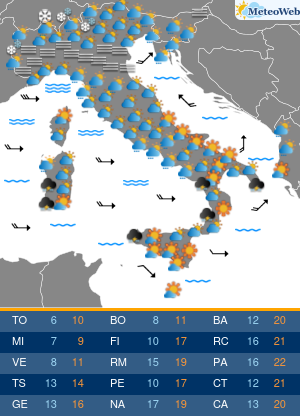
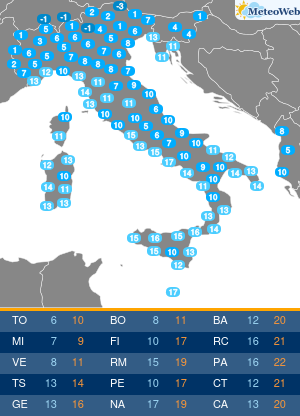
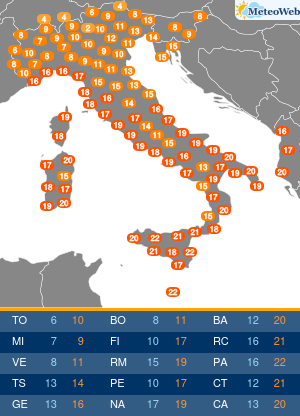
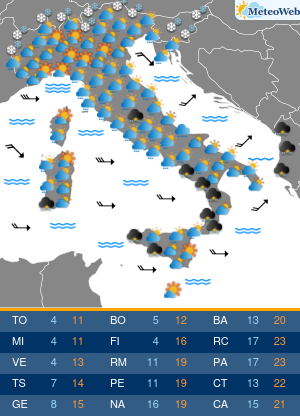
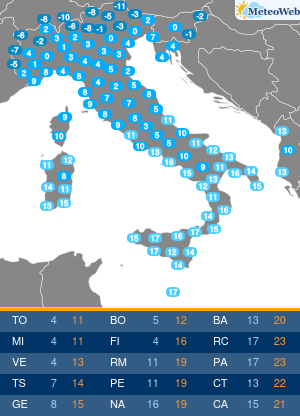
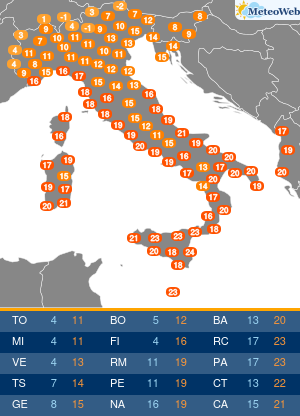
Un team di ricercatori, diretti da Graziano Pesole, dell’Istituto di biomembrane, bioenergetica e biotecnologie molecolari del Consiglio nazionale delle ricerche (Cnr-Ibiom) di Bari, e dell’Università degli Studi di Bari Aldo Moro, ha messo a punto una metodologia innovativa basata sull’utilizzo di una tecnica avanzata di biologia molecolare denominata “droplet digital PCR”, che è in grado di quantificare in modo estremamente accurato la carica batterica presente in un campione, riporta un comunicato stampa del Cnr. La tecnica si basa sulla quantificazione del numero assoluto di copie del gene 16S rRNA, presente nelle cellule batteriche, tenendo in considerazione anche lo stato di degradazione del DNA. Il metodo è stato pubblicato su Microbial Genomics.
“Anche se non li vediamo, gli organismi microbici sono le forme di vita più diffuse e abbondanti sulla terra e hanno un profondo impatto in moltissimi ambiti, dal controllo dell’equilibrio degli ecosistemi marino e terrestre fino a molti aspetti della fisiopatologia degli organismi pluricellulari, incluso l’uomo”, spiega Pesole. “Il microbioma umano, in particolare quello intestinale, è essenziale per la nostra salute: facilita la digestione delle sostanze nutritive, sintetizza vitamine, degrada sostanze tossiche e coadiuva lo sviluppo del nostro sistema immunitario”.
 Lo studio del microbioma ha visto uno straordinario sviluppo negli ultimi anni, grazie all’avvento delle piattaforme di sequenziamento massivo del DNA e alla conseguente messa a punto di tecniche che, in modo semplice e relativamente economico, consentono di determinare in termini percentuali la composizione delle specie microbiche di una grande varietà di campioni. “Tuttavia, per comprendere l’impatto funzionale della componente microbica degli organismi, per gli aspetti sia fisiologici che patologici, è importante ottenere una quantificazione assoluta e non relativa della carica microbica. Il nostro studio evidenzia che la valutazione preliminare dell’integrità del DNA è fondamentale per l’affidabilità delle differenze di carica microbica osservate tra campioni diversi e propone una correzione da applicare per i campioni di DNA metagenomici degradati”, conclude Pesole. “Questa nuova metodologia, semplice e accurata, potrà avere un vasto campo applicativo e consentirà di approfondire in modo significativo l’impatto funzionale del microbioma, in condizioni sia fisiologiche che in quelle di disbiosi, associate a moltissime patologie quali l’obesità, il diabete, varie malattie autoimmuni e forme di tumore”.
Lo studio del microbioma ha visto uno straordinario sviluppo negli ultimi anni, grazie all’avvento delle piattaforme di sequenziamento massivo del DNA e alla conseguente messa a punto di tecniche che, in modo semplice e relativamente economico, consentono di determinare in termini percentuali la composizione delle specie microbiche di una grande varietà di campioni. “Tuttavia, per comprendere l’impatto funzionale della componente microbica degli organismi, per gli aspetti sia fisiologici che patologici, è importante ottenere una quantificazione assoluta e non relativa della carica microbica. Il nostro studio evidenzia che la valutazione preliminare dell’integrità del DNA è fondamentale per l’affidabilità delle differenze di carica microbica osservate tra campioni diversi e propone una correzione da applicare per i campioni di DNA metagenomici degradati”, conclude Pesole. “Questa nuova metodologia, semplice e accurata, potrà avere un vasto campo applicativo e consentirà di approfondire in modo significativo l’impatto funzionale del microbioma, in condizioni sia fisiologiche che in quelle di disbiosi, associate a moltissime patologie quali l’obesità, il diabete, varie malattie autoimmuni e forme di tumore”.